はじめに
新型コロナウイルス感染症によって引き起こされた世紀に一度の壊滅的パンデミックから、世界規模での画期的なワクチン開発が始まった。このような規模の取り組みは現代医学史上、例を見ない。2020年6月には133種類ものワクチン候補(うち10種類は臨床試験中)が世界保健機構(WHO)のリストに記載され、目下、新型コロナウイルス感染症の病原体であるSARS-CoV-2に対する試験が進んでいる1。武漢で最初にウイルスが分離されてから、わずか数か月という極めて短期間のうちに、これ程の段階に達したのは2ワクチン研究では前代未聞の話だ。例えば、ジカウイルスやRS(呼吸器合胞体)ウイルスの場合、70年間も研究が行われているのに、認可されたワクチンは存在しない。科学者たちも十分自覚している通り、エイズやデング熱などの感染症の病原体に有効なワクチンを見つけることは至難の業だ。つまり現実的には、新型コロナウイルス感染症のワクチンが認可されて利用可能となるまでに、現在の世界的な努力が拍車をかけたとしても、1年はかかる3。それでも、大半の専門家は、集団免疫を獲得し、感染再発の波を抑えるには、安全で有効なワクチンが最も手頃で長期的な解決策であることを認めている4。幸運にも、ワクチン開発が有利なスタートを切ったのは、SARS-CoV-2と同じ科に属し、2003年SARSパンデミックの病原体であるSARS-CoV-1や、2012年の中東呼吸器症候群流行の病原体であるMERS-CoVなどのウイルス研究があったおかげだ。これらの研究から、ウイルス表面の王冠状の突起、いわゆる、スパイクタンパクが有効な抗体を誘導する可能性が高いという意見の一致が得られた5–7。
だが、1つのワクチンが新型コロナウイルス感染症の全症例に有効かつ適用可能なのか、という大きな懸念がある。現在のワクチン開発の取り組みは、中国やアメリカ、ヨーロッパに集中している。これらの地域で製造されたワクチンはどれも、他の地域で利用できるのだろうか。この疑問がより現実味を帯びてくるのが、開発途上国、特に東南アジアの開発途上国である。同地域の医学研究は予算や設備も限られている。ワクチンの研究開発(R&D)を行うには莫大な費用がかかるし、そのような制約された環境では、たとえ実りある成果が得られたとしても時間がかかるだろう。このため、他国の進捗状況を無視してでも、国内の新型コロナウイルスワクチンの研究に専念せねば、とのプレッシャーが高じている。東南アジアの政府指導者が8,9そのような発言をしているのは無理もない。ここで気がかりな点は、「外国の」ワクチンが使い物にならぬほど、このウイルスが国によって異なるのかということだ。また、ウイルスの急速な進化により、ワクチンへの耐性が生じたり、時間の経過と共にその危険性が増すようなことはないのだろうか。これらの疑問に答えるには、SARS-CoV-2のゲノムを調べ、ウイルス突然変異の本質を理解する必要がある。このため本稿では具体的にSARS-CoV-2の変異速度や、世界、特に東南アジア諸国におけるウイルスのゲノム分布、そして今後のワクチンの行方について幅広く論じる。
SARS-CoV-2の変異速度について
まず突然変異とは、遺伝子構造が変化することだ。遺伝子は4つの文字列(A、C、T、Gの塩基配列)として捉えられるが、ウイルスという生命体は極めて原始的で、これらの文字がエンベロープ[厳密にはカプシド(タンパク質の外殻)]に包まれているだけの存在だ。ウイルスはこれらの文字を宿主の細胞内でコピー・アンド・ペーストして複製するが、このコピー機能が正確性に欠けるため、これらの文字は時間の経過と共におのずと変化する。これが突然変異である。ここで重要な点は、突然変異は有効なワクチン製造の可否を決めるウイルスの特性の一つと考えられることだ。実際、HIVとC型肝炎ウイルス(HCV)のワクチン開発の難しさは有名で、この2種類のRNAウイルスは、大半のウイルスよりも早い速度で変異を起こす。高い変異率は遺伝的に多様なウイルス集団[厳密には類似種(quasispecies)という]を作り出し、地域分布が異なる様々な亜型を生み出す。例えば、HIVとHCVには、それぞれ、9種類と7種類の亜型があり、これらの亜型間で無数の組み換えが生じている。HIV-01AEとHCV-1bは、現在、東南アジアで最も多く見られるタイプだ。インフルエンザウイルスのRNAと亜型は急速に変化するので、これらのワクチンのデザインは毎年更新する必要がある。もう一つのRNAウイルスであるデングウイルス(DENV)の変異から生じた4種の血清型は、常に東南アジアを襲っているものだ。遺伝的にはHIVほど多様でないにせよ、この4種のDENVを十分にカバーするワクチン開発は骨の折れる仕事である。
RNAウイルスは急速に変異を起こすウイルスの代表格だが、その理由はRNAウイルスがゲノムをコピーする際に、ミスを起こす傾向が高いためだ。だが、ウイルス自体にとって、突然変異は両刃の剣である。変異はウイルスを新たな細胞環境に適応させ、宿主の免疫反応から逃れるのを助けることもあれば、ウイルスに害を与え、その複製能力を弱めることもある。突然変異の大半は中立的なもので、ウイルスタンパクを一切変化させない。この変異がもたらしている影響力のバランスが、ウイルスの運命を左右する。例えばポリオを考えてみよう。ポリオウイルスの変異速度はHIV-1よりも早い。この変異はポリオウイルスが環境に適応することが極めて難しいという制約下で生じている。つまり、その子孫ウイルスの多くが変異後に死滅してしまうため、ポリオウイルスの変異耐性は、HIV-1よりもずっと低くなるということだ10。今日、ポリオはワクチンによって実質的に根絶されている。奇妙なことではあるが、実はウイルスの変異はワクチン製造の役に立つ。ヒトにとって危険なウイルスは、動物細胞などヒト以外の宿主の中で十分な長期間培養された場合、生まれてくる子孫ウイルスは何度も変異を起こし、ヒトへの病原性を喪失する。この革新的な手法によって、麻疹やおたふく風邪のウイルスなどに有効な弱毒性の生ワクチン(LAV)が生み出された11。つまり、変異を盛んに起こすウイルスについて、より危険だとか、ワクチン耐性があるなどと言うのは間違っているが12、突然変異がRNAウイルスをさらに予測不可能なものにすることは、あながち間違いとは言えない。
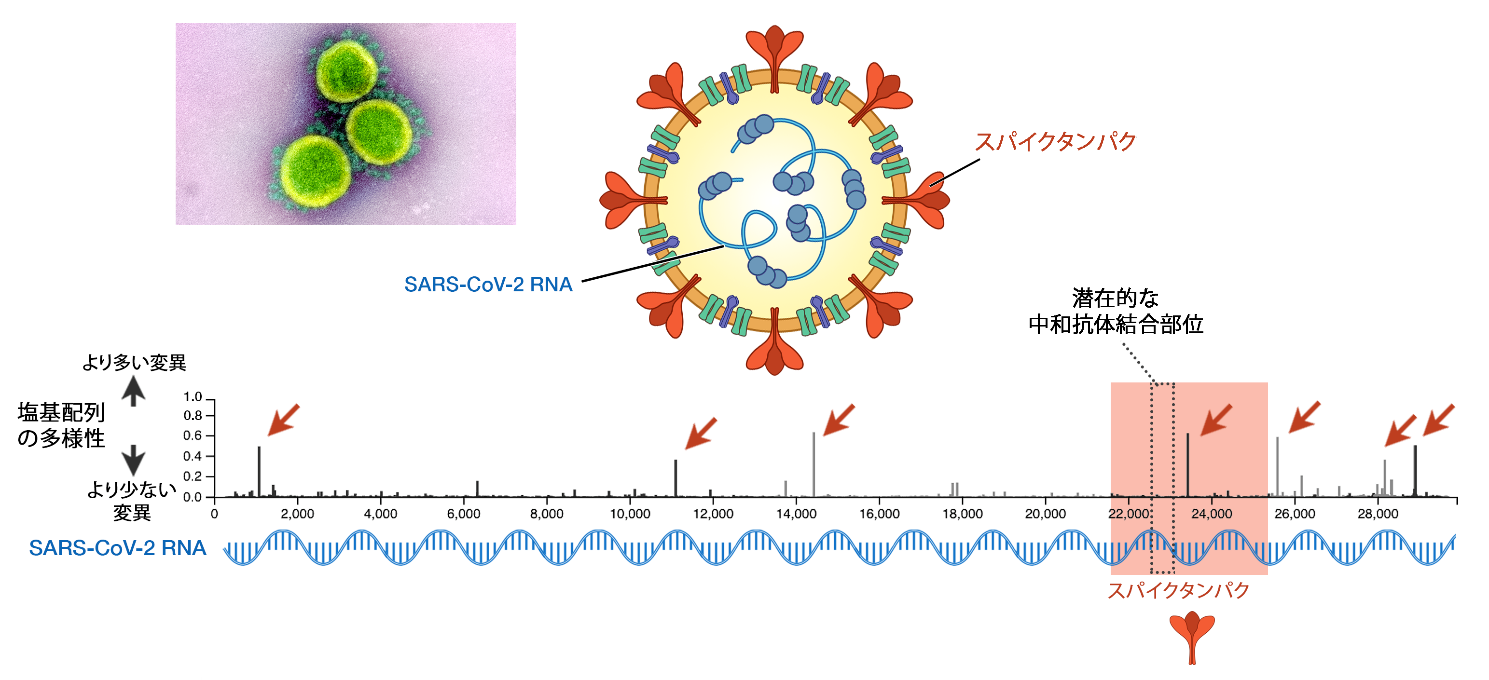
図1 SARS-CoV-2とそのゲノムの多様性。塩基配列の部位はⅩ軸で示し、塩基配列の多様性はY軸で示す。スパイクタンパクをコード化する遺伝子は赤く塗って強調してある。多様性の値が高いことは、その箇所での変異が多いことを表す。赤矢印で指した7箇所は多様性の値が高い箇所で、変異が発生しやすい箇所(mutational hotspots)と呼ばれる。そのうちの一つ(塩基配列23,403番の部位)は、スパイクタンパクをコード化する遺伝子の中に位置するが、点線の囲み(22,559番から23,143番の部位)で示された潜在的な中和抗体結合部位の外に位置する。データの提供元は2020年5月29日に更新されたGISAIDライブラリー13。電子顕微鏡写真の提供元はNIAID。
SARS-CoV-2自体がRNAウイルスであるため、当初はこのウイルスも予測不可能な変異特性を示すのではないかと懸念されていた。そこでゲノムデータが収集され、ウイルスRNAの特徴が次々に解明されるに従って、どうやら変異は十分に抑制されており、変異がウイルスの重大な変化を全て停止させるらしいということも分かってきた。これは安心材料と言える。SARS-CoV-2の世界的なゲノムデータは、GISAIDのリポジトリ(https://www.gisaid.org)から全て入手できるが、現在のところ、このゲノムにはほとんど多様性が見られない(図1)13。ゲノム全体の中で、大きな多様性を示しているのはわずかに7箇所だが、このゲノム全体は塩基配列30,000個近くに及ぶ長さを持ち、これまでに知られているRNAウイルスの中でも最長の類だ。これを他のウイルスと比較すると、どのくらい多様なのだろうか?SARS-CoV-2とHIV-1の進化系統樹を並べて、その違いを説明してみよう(図2)14。変異速度が最も早い生命体の一つであるHIV-1と比べるのは少々不公平だが、確実な点は、SARS-CoV-2のゲノムの全ての分離株が十分に保存されており、これらにHIV-1と同様の中和抗体結合部位がスパイクタンパクの中にあるのが共通していることだ5,15。さらに、現在進行中のパンデミックでは、ウイルスのヒト宿主への適応性を示す証拠が乏しいことから、SARS-CoV-2のワクチンは、流行中の全てのウイルス株をカバーするものと思われる14。
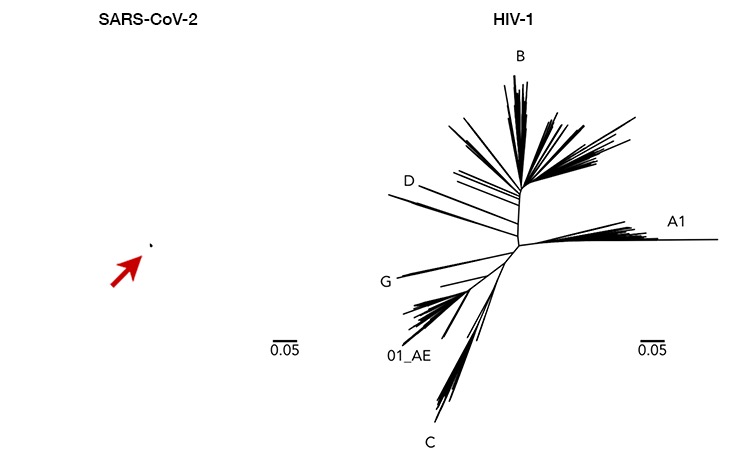
図2 この図はSARS-CoV-2(左、矢印が指す点)とHIV-1(右)の遺伝子の多様性の比較を示す。線が長いほど、2つの株同士の類縁は遠い、すなわち、より多く変異したゲノムということだ。この同尺度による並置は、今のところSARS-CoV-2の多様性がいかに微々たるものであるかを示す。HIV-1の全ての変種をカバーするには、非常に幅のあるワクチンが必要だが、SARS-CoV-2の全ての種類は1つのワクチンでカバーできそうだ。とは言え、SARS-CoV-2とHIV-1が異なる(例えば、感染や宿主の病変形成などの)特性を持つ、全く別のウイルスであることは指摘しておかなければならない。本図の使用にはモルガン・ローランド(Morgane Rolland)博士の許可を得ている14。
SARS-CoV-2の進化が、まだ序章に過ぎないとしても、これまでの分子時計の計算から、このウイルスの変異速度が極めて緩やかであることが明らかになっている。GISAIDが精選した5,000株以上のゲノム分離株を考慮すると、SARS-CoV-2の変異速度は8×10-4塩基置換(s)/塩基(n)/年(y)と推定される16。つまり、全ての塩基(AからU、CからGなど)には、1万分の8で変異の確率があるということだ。1年間の流行の後には、ゲノム全体に約25塩基の置換が見込まれる。比較のために挙げると、季節性インフルエンザウイルスは、少なくとも1年間に50塩基の置換を起こすが、そのゲノムサイズはSARS-CoV-2の半分しかない17。SARS-CoV-2の変異速度は、強毒性(SARS-CoV-1やMERS-CoV)や風邪をひきおこすような弱毒性(例えばHCoV-OC43)の他のヒトコロナウイルス(CoVs)の変異速度と同程度だが、他のRNAウイルスに比べるとずっと遅い(図3)。RNAウイルスの進化速度は、DNAウイルス(例えば単純ヘルペスウイルスなど)よりもずっと早い一方、コロナウイルスの進化速度はRNAウイルスの中では遅く、ゲノム安定性とワクチンによる予防効果が極めて高いRNAウイルスとして知られている麻疹ウイルスと同程度である。コロナウイルスの進化速度が遅い主な理由は、そのゲノムサイズの大きさ、そして複製時のRNA合成における読み取りミスを防ぐユニークなRNAの校正機能にある18。
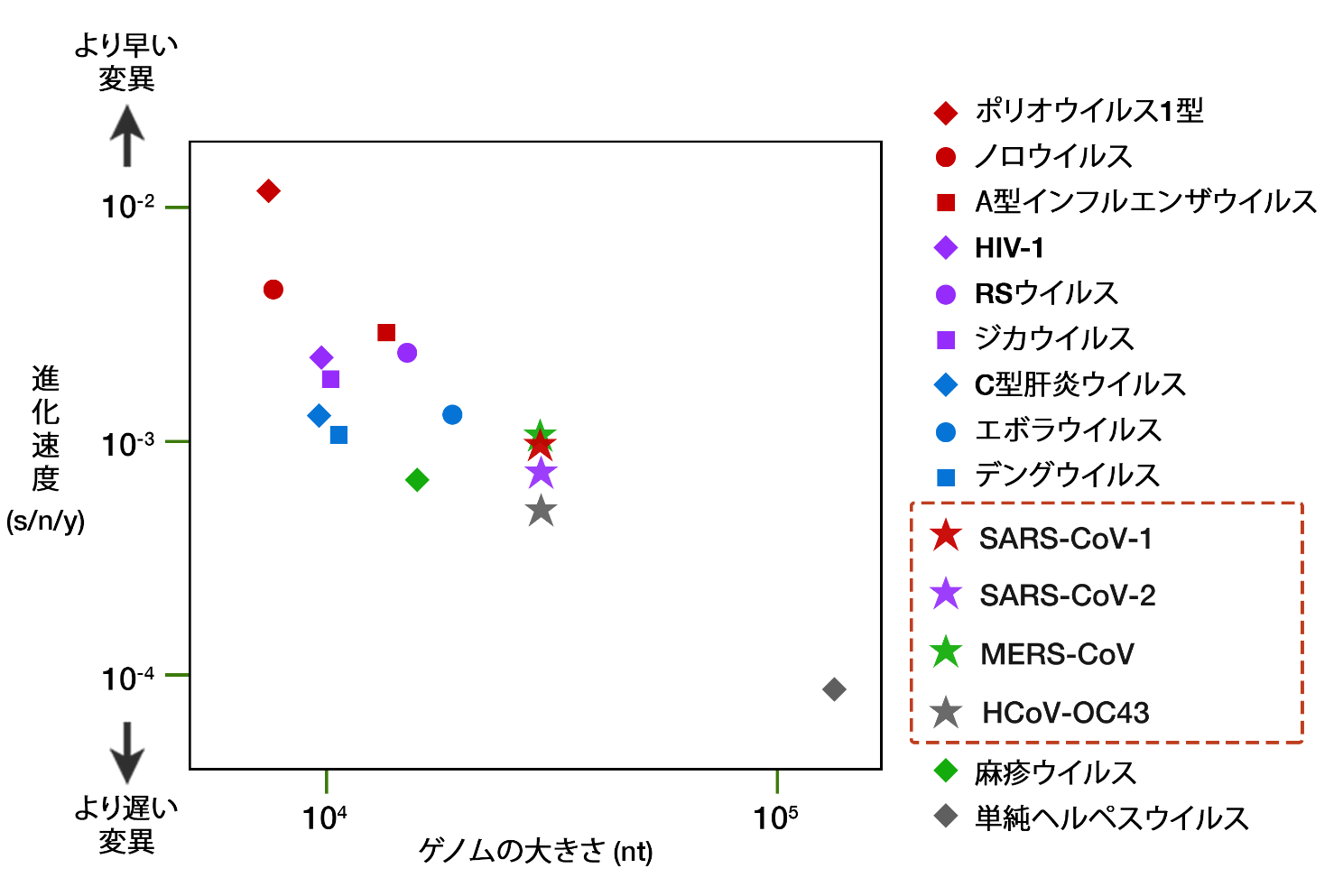
図3 RNAウイルスの進化速度の比較。進化速度(s/n/y、塩基置換を塩基あたり、年あたりで示す)はy軸上、ゲノムサイズ(nt、塩基数に基づく)はx軸上に示す。より大きなゲノムを持つ生命体は、変異速度がより遅い傾向にある。SARS-CoV-2を含むコロナウイルス科は、30,000ntという巨大なゲノムを持ち、その他のRNAウイルスに比べて遅い変異を示す。DNAウイルスの単純ヘルペスウイルスは、比較のために示すもの。データは複数の資料を基に編集した16,17,19–22。
SARS-CoV-2の変異の兆候を見ると、今のところ、この変異がワクチンの結合を妨げる様子は無さそうだが、変異の危険性は軽視するべきでない。最近の報告の指摘によると、変異にはウイルスの病原性、つまり、病気を引き起こす力を変化させる可能性がある。スパイクタンパクでの変異(図1の23,403番の部位で、D614G変異とも呼ぶ)は、ウイルスが細胞に侵入する際に用いるヒト側の受容体、ヒトACE-2への結合性を強化する可能性がある23。もう一つの変異に、ウイルスがRNAをコピーするのに用いるポリメラーゼ機構に生じるもの(14,409番の部位、P323L変異)がある。ポリメラーゼは新型コロナウイルス感染症の治療に、現在認可されている唯一の抗ウイルス薬、レムデシビルの攻撃対象だ24。この2種の変異が、実際にウイルスの致死性や薬剤耐性の引き金となるかどうかについては、今後の研究が待たれる。ひとつ憂慮すべき点を警告しておくと、これらの変異ウイルスは、その他の多くの国々に広がって優勢型となる前に、ヨーロッパの致死率(CFR)の高い地域でまん延し始めていることである25。これらの変異株は、中国での大流行初期には見られなかったものだ。現に、これまでのSARS-CoV-2の変異株から、ウイルスの進化だけではなく、新型コロナウイルス感染症の全世界における感染の地理的パターンも浮かび上がってきた。以下ではこの系統発生学的研究について述べよう。
新型コロナウイルス感染症の世界的な感染パターン──5つの系統群
ウイルスゲノムを配列比較することで、2つの異なる地域で流行する2つのウイルス株間の近縁性を測定し、これらを共通の祖先に結びつけることができる。進化系統樹を作成することによって、どの株が最初に出現したのかを知ることができる。さらに、地理的な起源を特定することで、ある株がどのようにして広がっていったかを知る手掛かりを得ることができる26。例えば、現在、東南アジアで広く分布しているHIV-1株(01AE亜型)は、アフリカ型の分離株と遺伝的に類似していることが1970年代に判明した。後になって、これは当時、中央アフリカからタイに移住してきた性産業の従事者を介して広まったことが分かった。変異をさらに遡ると、HIV-1の祖先株(図2の中心点)は、1920年代のキンシャサでチンパンジーからヒトへと感染したものであることが判明した。ウイルス変異をさらに古い時代に遡ることも可能だ。HCVの共通祖先は紀元前11世紀に遡るもので、おそらく東南アジア起源であると解明されており、B型肝炎ウイルス(HBV)は、人類が先史時代に旧世界から移動したことで拡散したものと考えられる。
またネクストレイン(Nextstrain、https://nextstrain.org/ncov/global)27には、これまでに収集された世界中のゲノムデータが見事に視覚化されており、これを使って変異をたどれば、SARS-CoV-2の共通祖先が出現したのは2019年12月1日であり、その地理的発生源は中国湖北省の武漢と推定することができる28。SARS-CoV-2ゲノムにおける、SARS-CoV-1ゲノムとの相同性は76%に過ぎないが、コウモリのコロナウイルスゲノムとの相同性は実に96%である。これはコウモリのコロナウイルスが、種間の類縁関係が最も近い祖先であることを示している29。コウモリとの違いはスパイクタンパクにあるが、このスパイク領域がSARS-CoV-2により近似しているのが、センザンコウのコロナウイルスだ30。このことから、SARS-CoV-2が自然宿主であるコウモリを飛び出し(コロナはコウモリに死をもたらすウイルスではない)、中間宿主と思われるセンザンコウに感染して(死なせ)、その後、さらに変異してヒトに感染したと推測できる31,32。武漢で2020年1月5日に採取されたウイルス(分離株名はWuhan/WH04/2020)は、コウモリの先祖株に最も近い系統で、B系統群と呼ばれることになった。だが、日付が最も古い2019年12月26日のSARS-CoV-2の標本(Wuhan-Hu-1/2019、現在はゲノム参照用に用いられるもの)には、2つの特徴的な変異(8,782番と28,144番の部位)があり、これが先祖株とこの系統との類縁関係をさらに遠くしている。この系統はヒトにおけるSARS-CoV-2の系統発生的起源を形成する型で、A系統群と呼ばれる。この武漢における初期の分岐から、AとBの両系統群が湖北省の外に持ち出され、その後、アジアと世界の各地に広がって行った。
SARS-CoV-2の系統をさらに遡って分類するため、数々の取り組みが行われている。その手法には議論があり、ある論文は分岐が3つの型に分かれる33と仮定し34、ある査読前論文には16もの型を定めるものもあった35。GISAIDは一時、「S」、「G」、「V」と「その他」という、図4の分岐図に見られる4つの系統群を設けていた。ランバウト(Rambaut)らが提案した可変的なシステム(センザンコウの系統、https://pangolin.docs.cog-uk.io)は、新規変異の度に新たな番号を割り当てるもので、厳格なシステムではあるが、結局、これらの識別番号は短命なものとなった37。「型」数の増加は、症例数が急増していては無理もないが、分類学上の名称を過度に追加しても、時が経てばその実用性は無くなるし、提唱された名称が様々になることは言うまでもない。残念ながら、ウイルスを種より下のレベルで分類する普遍的な方法は存在しない。例えば、HIV-1の「亜型」は、インフルエンザウイルスの「亜型」とは、事実上の階層関係にはない。このため、SARS-CoV-2については、「年次系統群(year-clade)」に基づいて命名するという点で意見が一致した38。系統群AとBは、それぞれに19A、19Bと呼ばれることになった。祖先株の19B系統群は、感染規模は小さかったが、2019年12月に最初に中国で広まり、ルーツとなる19A系統群は、主に中国とその他のアジアの全域に広まった。スパイク領域に変異(D614G)が生じたのは19A系統群で、これは2020年2月のヨーロッパでの大流行の際に生じ、20A系統群を生み出した。この20A系統群が急速に大陸全土に広まり、現在の世界的流行で多数を占める系統群となった。この時から新たな系統群を指定するには、世界の罹患率のうち20%以上に達していることが条件となった。このようにして、20A系統群は2月後半に分岐し、ヨーロッパ(ベルギー、スウェーデン)では20B系統群に、北米では20C系統群となった37,39。そのようなわけで、これまでに計5種の系統群の存在が確認されている。ネクストレインでは、この命名法を現在使用しており(図5を参照)、また今後も使用していく見込みである。
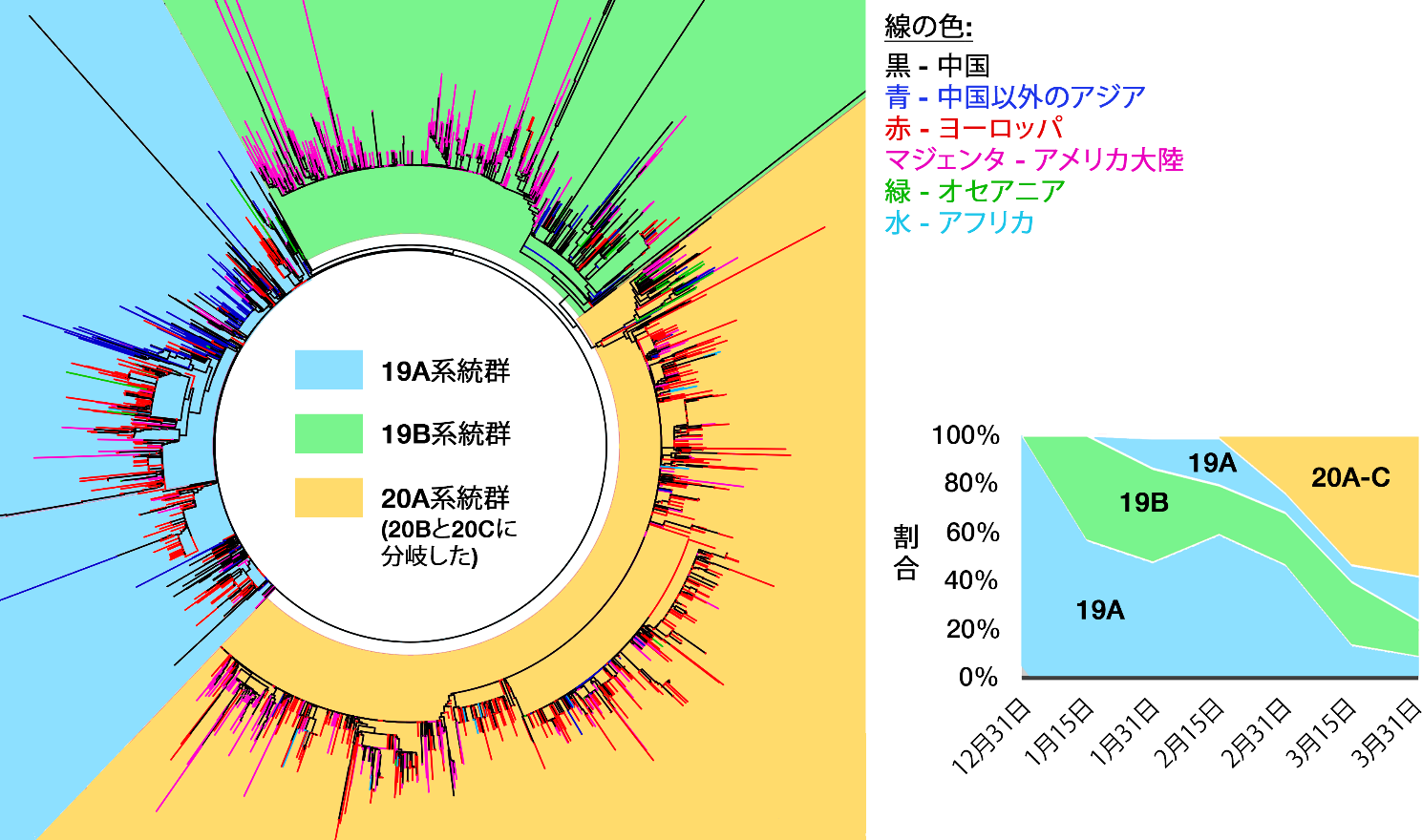
図4 SARS-CoV-2のゲノム疫学。本図は2020年5月12日のGISAID報告書に基づくが、系統群の表示はネクストレインの新たな「年次系統群」の分類に基づいて改定してある36。系統群の定義や、地理的詳細については本文を参照。挿入図 2月後半に出現した20A系統群(とその分枝の20Bと20C)は、徐々に以前の系統群に取って代わりつつある。
ウイルス集団が混在する地域(中国など)から出たウイルス分離株の一つが、偶然にも新しい、感染の土壌となる地域(ヨーロッパなど)にもたらされると、その特定の分離株は優勢な株となって広がり、元の集団とは異なるウイルス集団が生じる。これは「創始者効果」と呼ばれ、国の間でSARS-CoV-2の様々な系統群が分布している背景として、これが原動力として存在する可能性が最も考えられる。そうだとしても、新型コロナウイルス感染症の罹患率と死亡率には大きな地域差がある。例えば、致死率は中国国内(5.6%)と比べると、中国国外では3倍は高い(15.2%)40。このため、SARS-CoV-2は湖北省の外に持ち出された後、淘汰圧によって盛んに変異を起こし、より毒性と病原性の強い変異株が生じたと推測できる。先ほど述べた、変異が発生しやすい箇所での変異は、実際の臨床検査で高い毒性と関連があるとされており、中でもD614Gのスパイク領域変異を有する20A系統群の世界的な罹患率は着実に増えつつある(図4の挿入図参照)41。ただし、感染者数と致死率に作用する要因は無数にあり、例えば、国ごとの年齢層の違い42、極端に異なる緩和政策の度合い、医療設備の格差43、そして人間集団に固有の遺伝的特徴の多様性も、初期の集団免疫に影響を及ぼすものとなる41,44。複雑な社会経済的側面については言うまでもないが、人種差別や経済格差のために、致死率は特定の集団に偏っている45,46。SARS-CoV-2のいずれか特定の変異が、実際にヒトに感染する際の毒性を強めるという決定的証拠が見つかるまで、20A系統群の著しい増加は、創始者効果の結果と考える他ない47。この仮説は、HIV-1やエボラウイルスなど、その他のウイルスの流行において証明済みだ。
東南アジアにおけるSARS-CoV-2の拡散
そのため、新型コロナウイルス感染症の東南アジアへの侵入と、初期の拡散を調査することで、創始者の系統群と、そこから生じたこの地域のSARS-CoV-2の系統の特性を明らかにすることに関心が集まっている。東南アジアで最初の感染者は1月13日にタイで報告された事例で、バンコクを訪れていた一人の武漢住民だった48。タイ国内での最初の感染者は1月31日に報告されたタイ人のタクシー運転手で、中国人乗客からの感染と判明した49。シンガポールで最初の感染者が出たのは1月23日で、同じく武漢から来た一人の旅行者が感染していた。市中で初のクラスター感染も、中国人団体旅行客が利用する地方商店から2月4日に報告された50。シンガポールにおける感染症の大流行は、その後、マレーシアへと広まった。マレーシアで最初の感染者はシンガポール経由でやって来た3人の中国人旅行者で、その報告は1月25日だった。マレーシア人感染者で最初の報告があったのは2月4日で、少し前にシンガポールに旅行していた51。その後、2月27日に大規模感染が生じたが、原因はクアラルンプールで行われたタブリーグ・ジャマート(Tablighi Jamaat)というイスラーム教徒の集会イベントだった52。この結果、3月9日にはこのイベントに参加したブルネイ人が、ブルネイ・ダルサラーム国で最初の感染者となった53。
ベトナム初の輸入感染事例は、1月22日に報告された武漢から来た父子だった。この事例報告は、その後間もなく、中国以外でヒトからヒトへの新型コロナウイルス感染を示す最初の科学的根拠として公表された54。ベトナム初の国内感染事例が発表されたのは2月1日で、輸入感染事例の父子との接触歴が確認された55。これに続く一連の感染者の大部分は武漢との関連が確認された。中国と地続きで国境を接するにもかかわらず、ベトナム国内では感染拡大が首尾よく回避され、発症率も抑えられている。低コスト型でも効果あるベトナムの緩和対策は、国際社会から称賛を受けた。また、ベトナムは2003年のSARS大流行の際にも上手く対応していた56。ベトナム人研究者は、多数の臨床標本を用いたウイルスRNAの塩基配列解析にも成功しており、これはGISAIDのゲノム・ライブラリーで見ることができる。
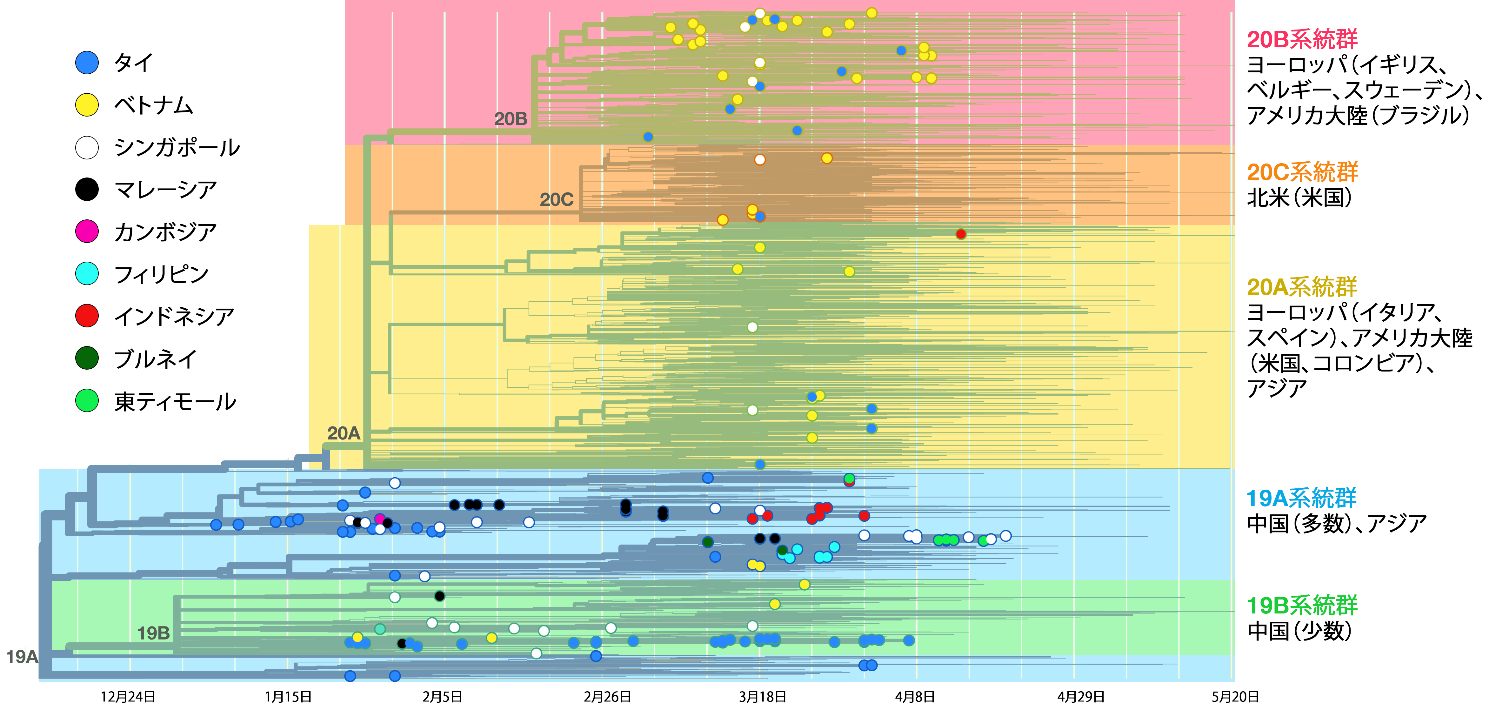
図5 SARS-Cov-2の完全分岐図、東南アジア諸国で採取された分離株は色付き点で示す。世界各地のゲノム分離株(合計標本数4,305株)を解析し、進化系統樹に配した。日付はx軸で示す。本系統樹は19A系統群(標準株Wuhan-Hu-1/2019)をルーツとする。湖北省で誕生した2つの系統群(19A、19B)を始めとするこのウイルスは、ヨーロッパとアメリカで3つの系統群(20A、20B、20C)に分岐した。系統群の東南アジアにおける拡散の詳細については本文を参照。データの提供元はネクストレイン、標本採取期間は2020年5月20日まで13。
2020年5月の時点で、GISAIDリポジトリはSARS-CoV-2の全ゲノム配列210例を東南アジア地域から入手したが、タイ、ベトナム、シンガポールからの提供が最も多かった。図5は最新の世界的な分岐図で、色付き点は東南アジアで採取された標本だ。図6は東南アジア各国のデータソースのおよその位置と、各国の系統群の分布を示す。タイとシンガポールの分離株の大半は、19Aと19Bの系統群に比較的等しく分岐しており、侵入経路が武漢であることを示している。初期のベトナムの標本も、この2つの中国系の系統群に由来するが、ベトナムやタイ、シンガポールでの最近の塩基配列解析の試みでは、20B系統群の分離株が無数に確認されている。これはやや懸念される、ヨーロッパから東南アジアへの感染者の入国の可能性を示すものだ。またマレーシアとブルネイの、これまでのほぼ全ての分離株が19A系統群と判明している。
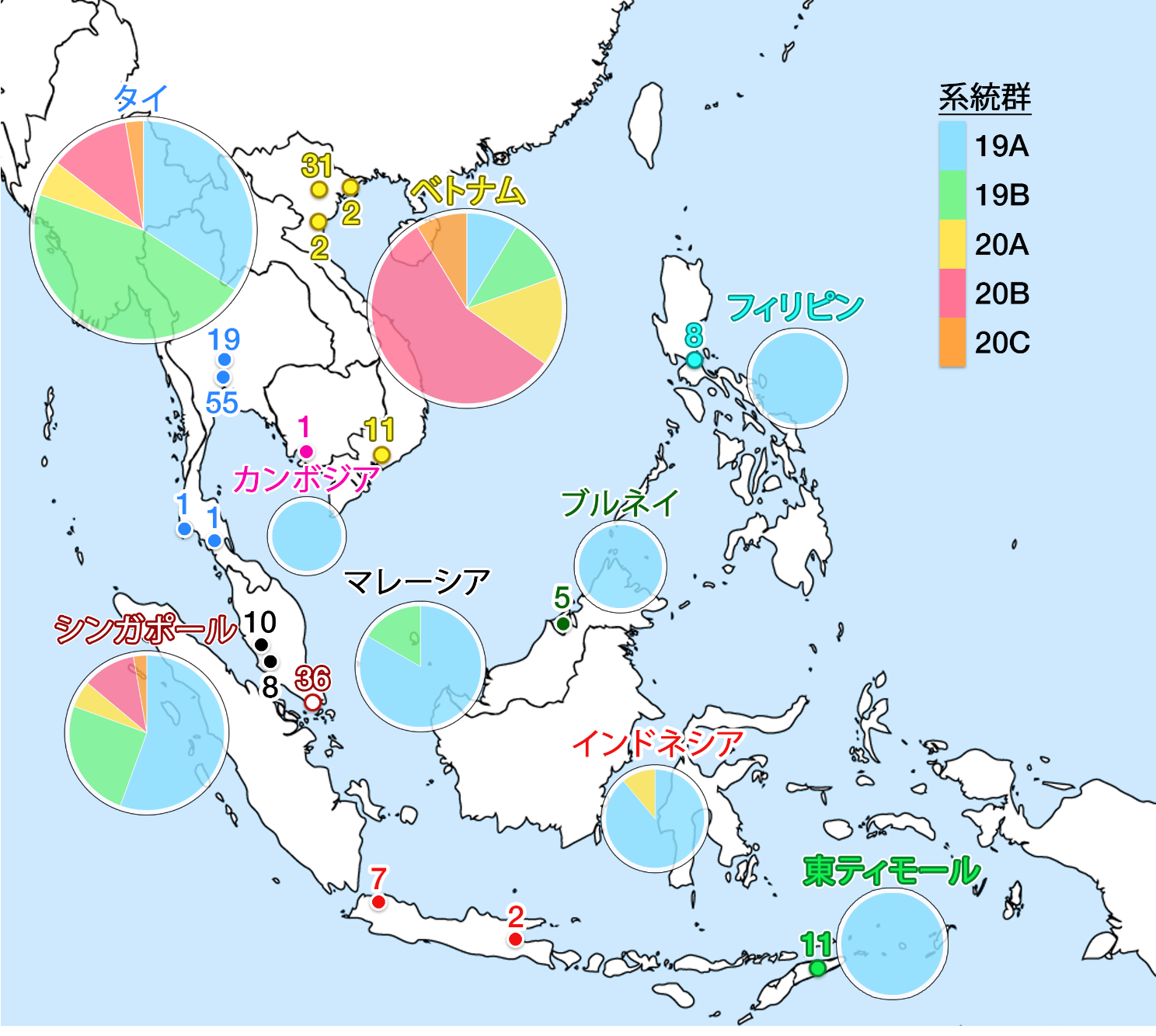
図6 この図は東南アジアにおけるSARS-CoV-2ゲノム株の分離場所と、各国での系統群の分布を示す。分離株の発生源と特定された地域は、地図上に色付き点で示す(点の色凡例については図5を参照)。点の上の数字は各地域で採取された標本の総量を示す。系統群の違いは国名の下の円グラフで示し、グラフの直径は採取された分離株の総量を比較表示したもの。19A系統群は、ほとんどの国で多数を占める系統群だが、タイやベトナム、シンガポールのより新しい分離株には、さらに多様な個体が見られ、ヨーロッパから感染者が入国した可能性がある。ミャンマーとラオスのデータは手に入らなかった。データの提供元は2020年6月4日に更新されたGISAIDライブラリーとネクストレイン13。
中国と東南アジア間の往来が絶えず頻繁であるために、特にSARS-CoV-2は中国との往来の多いタイとシンガポールから主に侵入してきたが、他の国々に関しては、この限りではない。カンボジアへの最初の輸入感染事例は1月27日に、一人の武漢住民から報告されたが57、カンボジアで最初の国内感染事例は、3月7日にシェムリアップで発表され、日本人との接触歴が確認された58。フィリピンで最初の事例は1月30日で、この感染者も武漢由来だったが、国内感染事例は3月6日に日本から帰国したフィリピン人の感染が報告されるまでは報告されていなかった。メトロマニラでの市中感染が確認されたのも、これと同じ日だ59。3月2日には2名のインドネシア人陽性者が、ジャカルタで確認され、日本人との接触歴があった。当初、インドネシアでは、輸入感染事例が発覚せず、国内第一報は他の東南アジア諸国に比べて遅れを取った。いずれにせよ、インドネシアにおける新型コロナウイルス感染症の罹患率は急上昇し、6月4日時点で28,818人に上った感染者数は、東南アジアではシンガポールに次ぐ2位で、その恐るべき致死率は6.1%と、他の全ての東南アジア諸国をはるかに上回り、アジアで5位となった60。しかしながら、インドネシアの検査率は1万人あたり1,225件と、世界でも最低レベルであり、この数字は大幅に低く見積もられている可能性がある61。最近では、感染発生の中心地が首都から東ジャワ州に移っている。原因はおそらくラマダーン終盤に帰省する人々の流れであると思われ、さらなる懸念が生じている。
カンボジア、フィリピン、インドネシアの分離株が1株を除き、すべて19A系統群(原型の武漢系統群)に分類されたことは、発生源となった国の一つである日本のゲノム疫学の結果とも合致している62。最新のインドネシアの分離株は20A系統群と特定され、ヨーロッパからの感染者入国の可能性がまたしても示された。このように、より新たなデータが大いに必要とされているのに、この3つの国の分離株の塩基配列解析の規模は広大な国土や多くの人口にも関わらず、東南アジアの三大国(タイ、ベトナム、シンガポール)にひどい遅れを取っている。インドネシアでの大流行はその後拡大し、3月21日には東ティモールで最初の市中感染を引き起こした63。東ティモールはすでに見事に分離株11株をGISAIDにアップロードしており、その全ての株が19A系統群だった。東南アジアで最後に新型コロナウイルス感染症の事例を報告した2国が、ミャンマーとラオスだ。ラオス人で1人目の感染者はタイでの集団発生からウイルスに感染し、これが3月24日に報告されている64。同じ日に、ミャンマーは国民2人が検査で陽性となったことを報告し、2人がそれぞれアメリカとイギリスから帰国しており、もう一つの感染者入国事例が示された65。この2つの国からの塩基配列解析の報告は、本稿の執筆時点では手に入れられなかった。
おわりに
ワクチン開発の成功は、病原性、ウイルスの適応度、多様性、そして進化動態というウイルスの4つの形質にかかっている。その高い病原性と適応度にもかかわらず、これまでのゲノム解析は、SARS-CoV-2は多様性が限られ、進化速度が遅いことを示している。だが、パンデミックは今なお進行中であり、データ収集は今後も継続していく必要がある。また、われわれは標本抽出バイアスに対しても批判的でなければならない。相対的に見ると、より多くのデータが北半球で作成されているからだ37。東南アジア諸国やその他の開発途上国のデータも同様に必要不可欠だ。また、ワクチンの研究開発が本質的にはウイルス進化に対する軍拡競争であったとしても、これを急ぐべきではない。効果のない危険なワクチンを時期尚早に展開しても、国民の信頼を損ねるばかりだ。不正確な情報、間違った懐疑論は、以前もポリオや麻疹ウイルスで見られたように、予防接種キャンペーンに長期的な損害を与える66。われわれは、コロナウイルス感染症再発の波を許すわけにもいかず、人々の3分の2が自然に免疫を獲得し、パンデミックが止むのを待っているわけにもゆかない67。ワクチンは、過去のパンデミックを制圧してきた。世界各地で一つのワクチンを使って新型コロナウイルス感染症を食い止められる可能性はあるのか。その答えは大いにある、と言うべきで、われわれはこのワクチン獲得のために、懸命な努力をする必要がある。
この記事を書くための有意義な議論や協力に対し、以下の方々に感謝の気持ちを捧げる。
林祥剛(Yoshitake Hayashi)教授、モルガン・ローランド(Morgane Rolland)博士、小瀧将裕(Tomohiro Kotaki)博士、ヤン・マルディアン(Yan Mardian)博士。
2020年8月6日 公開 (2020年6月5日 脱稿)
翻訳 吉田千春、監訳 山崎渉(東南アジア地域研究研究所)
参考文献
- 1 World Health Organization (WHO). 2 June, 2020. Draft landscape of COVID-19 candidate vaccines. https://www.who.int/who-documents-detail/draft-landscape-of-covid-19-candidate-vaccines. (Accessed 4 June, 2020).
- 2 World Health Organization (WHO). 20 January, 2020. Novel coronavirus (2019-nCoV) situation report-1. https://www.who.int/docs/default-source/coronaviruse/situation-reports/20200121-sitrep-1-2019-ncov.pdf?sfvrsn=20a99c10_4. (Accessed 25 May, 2020).
- 3 Anonymous. 2020. A COVID-19 vaccine might be ready within 18 months. But what happens then? Bill & Melinda Gates Foundation. https://www.gatesfoundation.org/TheOptimist/Articles/coronavirus-vaccine-development-gavi. (Accessed 26 May, 2020).
- 4 Burton DR, Walker LM. 22, April, 2020. Rational vaccine design in the time of COVID-19. Cell Host Microbe. 2020;27(5):695-698. doi:10.1016/j.chom.
- 5 Lan J, Ge J, Yu J, et al. 2020. Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor. Nature. 581(7807):215-220. doi:10.1038/s41586-020-2180-5
- 6 Smith TRF, Patel A, Ramos S, et al. 2020. Immunogenicity of a DNA vaccine candidate for COVID-19. Nat Commun. 11(1):2601. doi:10.1038/s41467-020-16505-0
- 7 Xie L, Sun C, Luo C, et al. February 2020. SARS-CoV-2 and SARS-CoV spike-RBD structure and receptor binding comparison and potential implications on neutralizing antibody and vaccine development. bioRxiv. 2020.02.16.951723. doi:10.1101/2020.02.16.951723
- 8 Bwhana PG. Jokowi: we need to develop our own COVID-19 vaccine. May 20, 2020. Tempo. https://en.tempo.co/read/1344441/jokowi-we-need-to-develop-our-own-covid-19-vaccine. (Accessed 4 June, 2020).
- 9 Kittisilpa J, Wongcha-um P. May 25, 2020. Thai researcher eyes affordable, accessible coronavirus vaccine for SE Asia. Reuters. https://www.reuters.com/article/us-health-coronavirus-thailand-vaccine/thai-researcher-eyes-affordable-accessible-coronavirus-vaccine-for-se-asia-idUSKBN2311DE. (Accessed 26 May, 2020).
- 10 Duffy S. 2018. Why are RNA virus mutation rates so damn high? PLoS Biol. 16(8):e3000003. doi:10.1371/journal.pbio.3000003
- 11 Hanley KA. 2011. The double-edged sword: how evolution can make or break a live-attenuated virus vaccine. Evol Educ Outreach. 4(4):635-643. doi:10.1007/s12052-011-0365-y
- 12 Fitzsimmons WJ, Woods RJ, McCrone JT, et al. 2018. A speed–fidelity trade-off determines the mutation rate and virulence of an RNA virus. PLoS Biol. 16(6):e2006459. doi:10.1371/journal.pbio.2006459
- 13 Nextstrain. Genomic epidemiology of novel coronavirus: global subsampling. Nextstrain. https://nextstrain.org/ncov/global. 30 May, 2020. (Accessed 4 June, 2020).
- 14 Dearlove B, Lewitus E, Bai H, et al. January, 2020. A SARS-CoV-2 vaccine candidate would likely match all currently circulating strains. bioRxiv. 2020.04.27.064774. doi:10.1101/2020.04.27.064774
- 15 Yuan M, Wu NC, Zhu X, et al. 2020. A highly conserved cryptic epitope in the receptor-binding domains of SARS-CoV-2 and SARS-CoV. Science. 368(6491):eabb7269. doi:10.1126/science.abb7269
- 16 Global Initiative on Sharing All Influenza Data (GISAID). 25 May, 2020. Genomic epidemiology of novel coronavirus: global subsampling. https://www.gisaid.org/epiflu-applications/next-hcov-19-app/. (Accessed 4 June, 2020).
- 17 Global Initiative on Sharing All Influenza Data (GISAID). 13 April, 2020. Real-time tracking of influenza A/H3N2 evolution using data from GISAID. https://nextstrain.org/flu/seasonal/h3n2/ha/2y?l=clock. (Accessed 31 May, 2020).
- 18 Surdel MC. 2014. Coronaviruses lacking exoribonuclease activity are susceptible to lethal mutagenesis: evidence for proofreading and potential therapeutics. PLoS Pathog. 10(7):e1003565. doi:10.1371/journal.ppat.1004342
- 19 Peck KM, Lauring AS. 2018. Complexities of viral mutation rates. Sullivan CS, ed. J Virol. 92(14):e01031-17. doi:10.1128/jvi.01031-17
- 20 Lau EHY, Hsiung CA, Cowling BJ, et al. 2010. A comparative epidemiologic analysis of SARS in Hong Kong, Beijing and Taiwan. BMC Infect Dis. 10:50. doi:10.1186/1471-2334-10-50
- 21 Cotten M, Watson SJ, Zumla AI, et al. 2014. Spread, circulation, and evolution of the Middle East respiratory syndrome coronavirus. MBio. 5(1):e01062-13. doi:10.1128/mBio.01062-13
- 22 Vijgen L, Keyaerts E, Moës E, et al. 2005. Complete Genomic Sequence of Human Coronavirus OC43: Molecular Clock Analysis Suggests a Relatively Recent Zoonotic Coronavirus Transmission Event. J Virol. 79(3):1595-1604. doi:10.1128/jvi.79.3.1595-1604.2005
- 23 Ou J, Zhou Z, Dai R, et al. March, 2020. Emergence of RBD mutations from circulating SARS-CoV-2 strains with enhanced structural stability and higher human ACE2 receptor affinity of the spike protein. bioRxiv. 2020.03.15.991844. doi:10.1101/2020.03.15.991844
- 24 Pachetti M, Marini B, Benedetti F, et al. 2020. Emerging SARS-CoV-2 mutation hot spots include a novel RNA-dependent-RNA polymerase variant. J Transl Med. 18(1):179. doi:10.1186/s12967-020-02344-6
- 25 Korber B, Fischer W, Gnanakaran SG, et al. January, 2020. Spike mutation pipeline reveals the emergence of a more transmissible form of SARS-CoV-2. bioRxiv. 2020.04.29.069054. doi:10.1101/2020.04.29.069054
- 26 Sagulenko P, Puller V, Neher RA. 2018. TreeTime: maximum-likelihood phylodynamic analysis. Virus Evol. 4(1):vex042-vex042. doi:10.1093/ve/vex042
- 27 Hadfield J, Megill C, Bell SM, et al. 2018. Nextstrain: real-time tracking of pathogen evolution. Bioinformatics. 34(23):4121-4123. doi:10.1093/bioinformatics/bty407
- 28 Lu J, Plessis L du, Liu Z, et al. April, 2020. Genomic epidemiology of SARS-CoV-2 in Guangdong province, China. Cell. S0092-8674(20)30486-4. doi:10.1016/j.cell.2020.04.023
- 29 Andersen KG, Rambaut A, Lipkin WI, Holmes EC, Garry RF. 2020. The proximal origin of SARS-CoV-2. Nat Med. 26(4):450-452. doi:10.1038/s41591-020-0820-9
- 30 Lam TT-Y, Shum MH-H, Zhu H-C, et al. 2020. Identifying SARS-CoV-2 related coronaviruses in Malayan pangolins. Nature. doi:10.1038/s41586-020-2169-0
- 31 Zhang YZ, Holmes EC. 2020. A genomic perspective on the origin and emergence of SARS-CoV-2. Cell. 181(2):223-227. doi:10.1016/j.cell.2020.03.035
- 32 Gao W-H, Lin X-D, Chen Y-M, et al. 2020. Newly identified viral genomes in pangolins with fatal disease. Virus Evol. 6(1):veaa020. doi:10.1093/ve/veaa020
- 33 Forster P, Forster L, Renfrew C, Forster M. 2020. Phylogenetic network analysis of SARS-CoV-2 genomes. Proc Natl Acad Sci. 117(17):9241 LP – 9243. doi:10.1073/pnas.2004999117
- 34 Mavian C, Pond SK, Marini S, et al. May, 2020. Sampling bias and incorrect rooting make phylogenetic network tracing of SARS-COV-2 infections unreliable. Proc Natl Acad Sci. 202007295. doi:10.1073/pnas.2007295117
- 35 Júnior IJM, Polveiro RC, Souza GM, Bortolin DI, Sassaki FT, Lima ATM. April, 2020. The global population of SARS-CoV-2 is composed of six major subtypes. bioRxiv. 2020.04.14.040782. doi:10.1101/2020.04.14.040782
- 36 Global Initiative on Sharing All Influenza Data (GISAID). 4 April, 2020. Full genome tree of all outbreak sequences. https://www.gisaid.org/hcov-19-analysis-update. (Accessed 29 May, 2020).
- 37 Rambaut A, Holmes EC, Hill V, et al. April, 2020. A dynamic nomenclature proposal for SARS-CoV-2 to assist genomic epidemiology. bioRxiv. 2020.04.17.046086. doi:10.1101/2020.04.17.046086
- 38 Hodcroft EB, Hadfield J, Neher RA, Bedford T. 3 June, 2020. Year-letter genetic clade naming for SARS-CoV-2 on nextstrain.org. Nextstrain. https://virological.org/t/year-letter-genetic-clade-naming-for-sars-cov-2-on-nextstain-org/498. (Accessed 4 June, 2020).
- 39 Eden JS, Rockett R, Carter I, et al. 2020. An emergent clade of SARS-CoV-2 linked to returned travellers from Iran. Virus Evol. 6(1):veaa027. doi:10.1093/ve/veaa027
- 40 Baud D, Qi X, Nielsen-Saines K, Musso D, Pomar L, Favre G. June, 2020. Real estimates of mortality following COVID-19 infection. Lancet Infect Dis. doi:10.1016/S1473-3099(20)30195-X
- 41 Bhattacharyya C, Das C, Ghosh A, et al. May, 2020. Global spread of SARS-CoV-2 subtype with spike protein mutation D614G is shaped by human genomic variations that regulate expression of TMPRSS2 and MX1 genes. bioRxiv. 2020.05.04.075911. doi:10.1101/2020.05.04.075911
- 42 Li H, Wang S, Zhong F, et al. February, 2020. Age-dependent risks of incidence and mortality of COVID-19 in Hubei province and other parts of China. medRxiv. 2020.02.25.20027672. doi:10.1101/2020.02.25.20027672
- 43 He Y. Illness and fatality risks of COVID-19 of general public in Hubei provinces and other parts of China. February, 2020. medRxiv. 2020.02.25.20027672. doi:10.1101/2020.02.25.20027672
- 44 Grifoni A, Weiskopf D, Ramirez SI, et al. May, 2020. Targets of T cell responses to SARS-CoV-2 coronavirus in humans with COVID-19 disease and unexposed individuals. Cell. doi:10.1016/j.cell.2020.05.015
- 45 Abedi V, Olulana O, Avula V, et al. 2020. Racial, economic and health inequality and COVID-19 infection in the United States. medRxiv. 2020.04.26.20079756. doi:10.1101/2020.04.26.20079756
- 46 Yancy CW. COVID-19 and African Americans. 15 April, 2020. JAMA. 323(19):1891-1892. doi:10.1001/jama.2020.6548
- 47 Farkas C, Fuentes-Villalobos F, Garrido JL, Haigh JJ, Barría MI. April, 2020. Insights on early mutational events in SARS-CoV-2 virus reveal founder effects across geographical regions. bioRxiv. 2020.04.09.034462. doi:10.1101/2020.04.09.034462
- 48 Cheung E. Wuhan pneumonia: Thailand confirms first case of virus outside China. 13 January, 2020. South China Morning Post. https://www.scmp.com/news/hong-kong/health-environment/article/3045902/wuhan-pneumonia-thailand-confirms-first-case. (Accessed 4 June, 2020).
- 49 Anonymous. 31 January, 2020. สธ.แถลง พบคนขับแท็กซี่ ติดไวรัสโคโรน่า เป็นคนไทยรายแรก ไม่มีประวัติไปจีน (MOPH announces taxi driver infected with coronavirus—first Thai case with no records of travelling to China). Thai Rath. https://www.thairath.co.th/news/local/bangkok/1761053. (Accessed 3 June, 2020).
- 50 Lien C-A, Khalik S. 4 February, 2020. Coronavirus: S’pore reports first cases of local transmission; 4 out of 6 new cases did not travel to China. The Straits Times. https://www.straitstimes.com/singapore/coronavirus-spore-reports-first-cases-of-local-transmission-4-out-of-6-new-cases-did-not. (Accessed 3 June, 2020).
- 51 Anonymous. 4 February, 2020. First case of Malaysian positive for coronavirus. Bernama. https://www.bernama.com/en/general/news_coronavirus.php?id=1811373. (Accessed 3 June, 2020).
- 52 Ananthalakshmi A, Sipalan J. 18 March, 2020. How mass pilgrimage at Malaysian mosque became coronavirus hotspot. Reuters. https://www.reuters.com/article/us-health-coronavirus-malaysia-mosque/how-mass-pilgrimage-at-malaysian-mosque-became-coronavirus-hotspot-idUSKBN2142S4. (Accessed 3 June, 2020).
- 53 Ministry of Health Brunei Darussalam. 9 March, 2020. Detection of the first case of COVID-19 infection in Brunei Darussalam. http://www.moh.gov.bn/Lists/Latest news/NewDispForm.aspx?ID=366. (Accessed 4 June, 2020).
- 54 Phan LT, Nguyen T V., Luong QC, et al. Importation and human-to-human transmission of a novel coronavirus in Vietnam. N Engl J Med. 2020;382(9):872-874. doi:10.1056/NEJMc2001272
- 55 Anonymous. 4 February 4, 2020.Bệnh nhân viêm phổi thứ ba xuất viện (Third pneumonia patient discharged). VnExpress. https://vnexpress.net/benh-nhan-viem-phoi-thu-ba-xuat-vien-4050123.html. (Accessed 4 June, 2020).
- 56 Reed J. 24, March, 2020. Vietnam’s coronavirus offensive wins praise for low-cost model. Financial Times. https://www.ft.com/content/0cc3c956-6cb2-11ea-89df-41bea055720b. (Accessed 4 June, 2020).
- 57 Sithirith M. 2020. COVID-19 in Cambodia: a double-edged sword and its triple effects on Democracy. CSEAS Newsl. 2020;(78).
- 58 Tostevin M, Williams A. 7 March, 2020.First Cambodian tests positive for coronavirus. Reuters. https://www.reuters.com/article/us-health-coronavirus-cambodia/first-cambodian-tests-positive-for-coronavirus-idUSKBN20U0HW. (Accessed 4 June, 2020).
- 59 Punzalan J. 6 March, 2020.Philippines’ new coronavirus cases now at 5, including potential local transmission. ABS-CBN News. https://news.abs-cbn.com/news/03/06/20/new-coronavirus-case-in-philippines-possible-local-transmission. (Accessed 4 June, 2020).
- 60 Gugus Tugas Percepatan Penanganan COVID-19. 2020. Peta Sebaran Kasus per Provinsi. Jakarta: Gugus Tugas Percepatan Penanganan COVID-19. https://covid19.go.id/peta-sebaran. (Accessed 4 June, 2020).
- 61 Anonymous. 2 June, 2020. Indonesia’s daily coronavirus testing consistently on target. Jakarta Globe. https://jakartaglobe.id/news/indonesias-daily-coronavirus-testing-consistently-on-target. (Accessed 4 June, 2020).
- 62 National Institute of Infectious Diseases. 2020. An Epidemiological Study of the SARS-CoV-2 Genome in Japan. Tokyo: NIID. https://www.niid.go.jp/niid/en/basic-science/467-genome/9598-genome-2020-1e.html. (Accessed 4 June, 2020).
- 63 Cruz N Da, Ungku F. 21 March, 2020. East Timor confirms first case of coronavirus: health ministry. Reuters. https://www.reuters.com/article/us-health-coronavirus-timor/east-timor-confirms-first-case-of-coronavirus-health-ministry-idUSKBN2180BI. (Accessed 4 June, 2020).
- 64 Wongcha-um P, Elgood G. 24 March, 2020. Laos records first two coronavirus cases: Thai media. Reuters. https://www.reuters.com/article/us-health-coronavirus-laos/laos-records-first-two-coronavirus-cases-thai-media-idUSKBN21B0X2. (Accessed 4 June, 2020).
- 65 Fullick N. 24 March, 2020. Myanmar reports first cases of coronavirus. Reuters. https://www.reuters.com/article/us-health-coronavirus-myanmar/myanmar-reports-first-cases-of-coronavirus-idUSKBN21B0HB. (Accessed 4 June, 2020).
- 66 Trogen B, Oshinsky D, Caplan A. May, 2020. Adverse consequences of rushing a SARS-CoV-2 vaccine: implications for public trust. JAMA. doi:10.1001/jama.2020.8917
- 67 Randolph HE, Barreiro LB. 2020. Herd immunity: understanding COVID-19. Immunity. 52(5):737-741. doi:10.1016/j.immuni.2020.04.012
筆者紹介
ユディル・オフィンニ(Youdiil Ophinni): 2011年にインドネシア大学で医学博士号を、2018年に神戸大学でウイルス学の博士号を取得。現在はマサチューセッツ総合病院(MGH)、マサチューセッツ工科大学(MIT)およびハーバード大学のラゴン研究所(the Ragon Institute of MGH, MIT and Harvard)でポストドクターを務める。
Citation
ユディル・オフィンニ(2020)「東南アジアにおける新型コロナウイルスの突然変異と拡散 ──今後のワクチンの行方──」CSEAS Newsletter 4: TBC.